采用CLC进行儿童痰液中微生物群落分析,比较了采用CLC 和 EBI Metagenomics工具的分析结果,表明CLC Bio MGM模块可以将所有的样本中菌群分类到门的水平;而EBI工具在15%的样本中都不能分配到门的水平。
前言:
本案例以儿童下呼吸道诱导痰为样本,通过V3-V4区的16SrRNA测序进行儿童下呼吸道微生物群落分析。测序数据用Biomedical Genomics Workbench及Microbial Genomics Module插件进行菌种的分类分析。结果发现口腔、咽喉、痰标本细菌群落分布相似,鼻、支气管的标本不同。鼻、口以放线菌为主,喉和痰以梭菌为主,支气管标本以蛋白杆菌为主。百分之四十一的支气管样本中未检出细菌。因此诱导痰可能不是所有个体下呼吸道微生物评价的有效方法。标准测试不能检测出支气管样本中的许多细菌类型,这表明我们对儿童下呼吸道菌群的了解仍不完善。
方法:
1、共招募二十名儿童,平均年龄10.7岁。诱导痰标本取自12名儿童,支气管刷洗来自其中14名,鼻、口、喉标本分别从15、16和17名儿童中取样。
2、样本经过处理后,进行可变区V3-V4的16S测序。
3、使用了配备Microbial Genomics Module(微生物插件)Biomedical Genomics Workbench (CLCbio,Qiagen)软件进行16S测序数据的处理。
4、微生物数据也用EBI Metagenomics工具进行处理,对比两个平台分析结果。
结果:
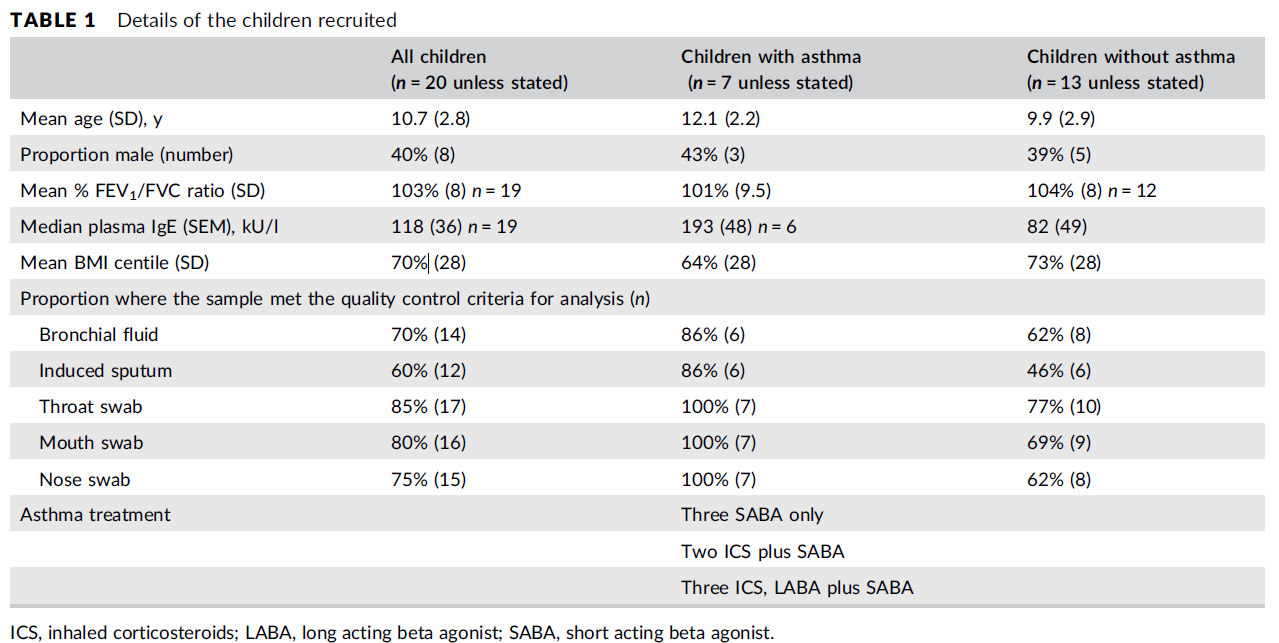
表一. 采样细节
 |
 |
|
|
|
|
|
|
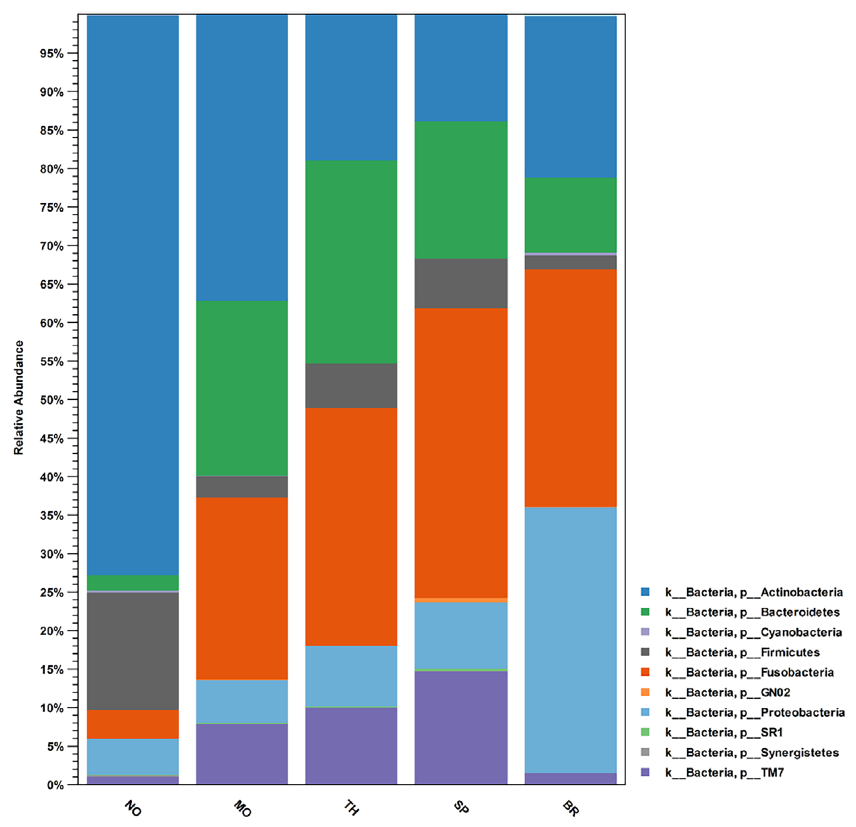
图一. 用CLCbio处理的儿童气管样本的OTU相对分布图(分类到细菌的门)。条形图表明每一种样本的分类组成。每个样品中细菌的详细摘要如图二所示。采样分组:NO,鼻子;MO,嘴;TH,喉咙;SP,痰;BR,支气管样本。 |
|
图二. 所有75个样本(即74个患者样本加上具有可检测细菌DNA的对照)的16S rRNA的OTU相对丰度(分类到细菌的门),描述了每个样品中的细菌的详细摘要。在水平轴上,P后面的数字是患者编号和采样分组:NO,鼻子;MO,嘴;TH,喉咙;SP,痰;BR,支气管样本。C2=检测细菌DNA的对照样品。 |
|
|
|
|
 |
|
 |
|
|
|
|
|
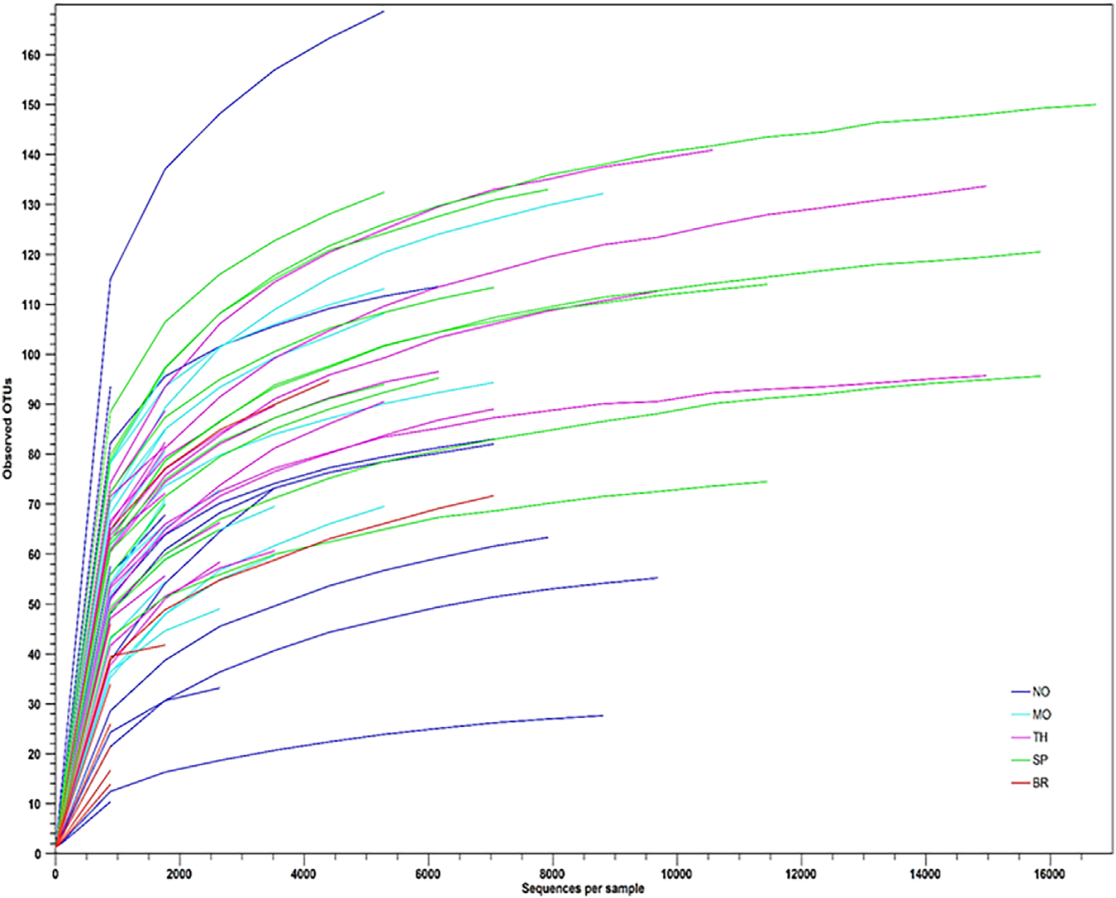
图三. 以样本分类OTU的alpha diversity多样性分析曲线图。不用样本以颜色区分:“鼻子”(NO),“嘴”(MO),“喉咙”(TH),“痰”(SP)和“支气管”(BR)。 |
|
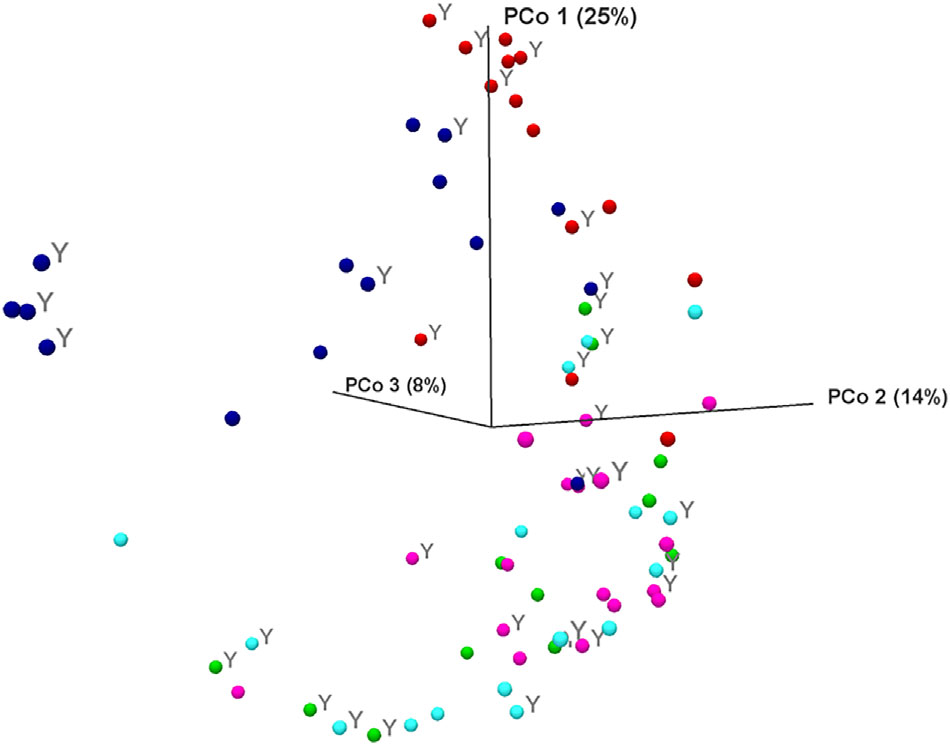
图四. 多样性分析表明,从哮喘和非哮喘儿童气道取样的细菌门群存在差异。基于DY0.5 UNIFRAC距离的所有样本主坐标(PCO)分析。颜色表示不同的样本队列 ( “鼻”(NO)、“嘴”(MO)、“喉”(TH)、“痰”(SP)和“支气管”(BR)样本)。Y=哮喘儿童样本 |
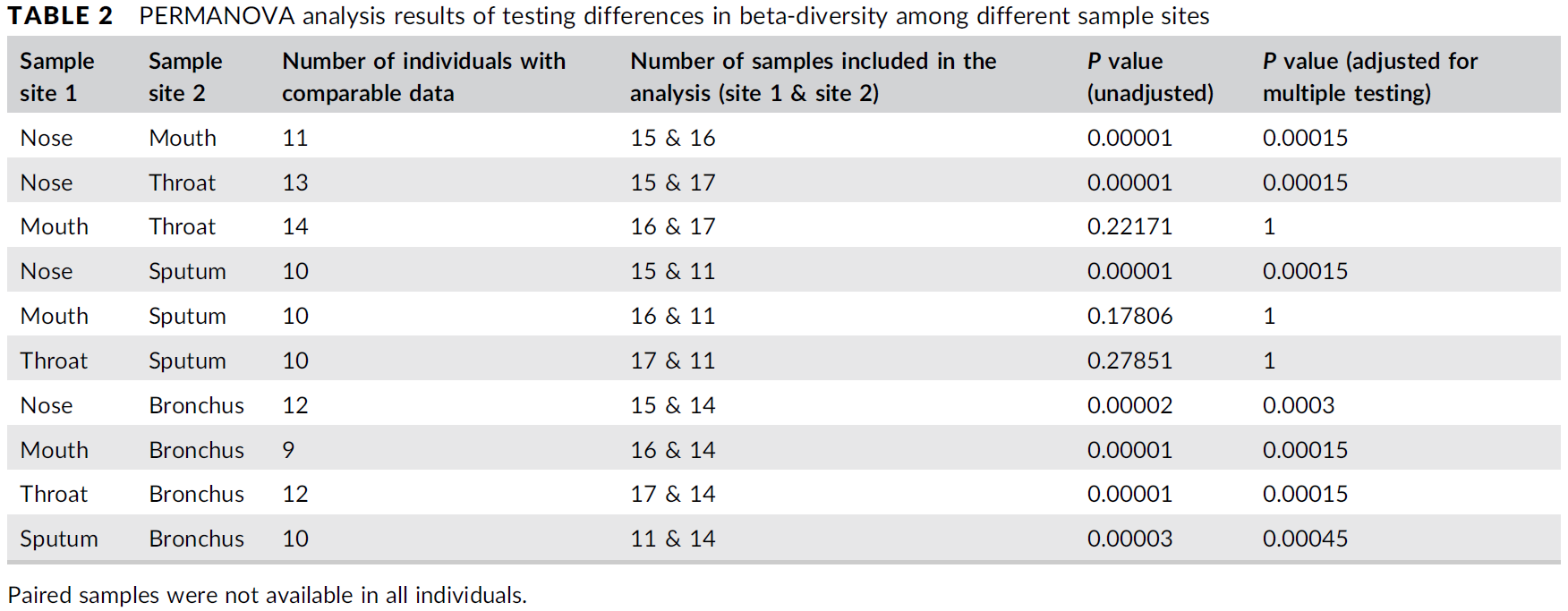
表二. 以UniFrac距离进行PERMANOVA进行Robust分析
结论:
1、文章结果比较了采用CLCbio 和 EBI Metagenomics工具的分析结果,结果表明CLC Bio MGM模块可以将所有的样本中菌群分类到门的水平;而EBI工具在15%的样本中都不能分配到门的水平。
2、添加微生物分析模块(MGM模块)的CLCbio为客户提供全视窗化宏基因组分析界面,提供优秀的算法和数据库用于分析微生物测序数据。
3、标准测试不能检测出支气管样本中的许多细菌类型,这表明我们对儿童下呼吸道菌群的了解仍不完善。
案例原文:
An S Q, Warris A, Turner S. Microbiome characteristics of induced sputum compared to bronchial fluid and upper airway samples[J]. Pediatr Pulmonol, 2018, 53(7).



