南非鲍(Haliotis midae)是一种腹足纲软体动物,分布地域广,从西海岸(萨尔达尼亚,Saldahna)到东海岸里特点(Riet Point),因此南非鲍生存环境的多样性导致了它遗传和基因表达的多样性,这是南非鲍能够在动态环境中生存的原因。最近,比较测序组装的研究表明454 reads与Illumina reads混合序列能够组装成高质量转录组。本文使用南非鲍RNA样本,比较常用的两个测序平台——Roche 454焦磷酸测序或Illumina SBS(Sequencing-by-synthesis,合成测序),在发现或预测适应多种环境的关键基因和微地理环境波动应激反应的关键基因方面优势。
实验设计、材料与方法:
1. 样本准备与组装
提取南非鲍mRNA样本,分别选择454测序与Illumina测序的样本,遵循各自操作规范,实验样本情况详见表1。使用Roche 454焦磷酸测序或Illumina SBS对之前的组织mRNA进行测序,获得全面的转录组。454焦磷酸测序在Roche 454 GSFLX平台进行,Illumina测序使用四色DNA SBS技术。使用CLC Genomics Workbench(8.5版)软件组装单一测序平台的测序结果与两个平台的混合测序结果。
表1 实验样本情况一览表

2. 原始数据、碱基对与组装
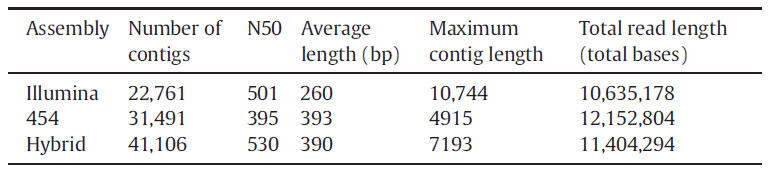
Illumina测序结果与454测序结果见表2。本文研究目的是为了展示,混合数据组装(两个测序平台的混合测序结果组装)与单一数据组装(单一测序平台的测序结果组装)相比的优势。大多数转录组组装的测评显示,混合数据组装得分更高,其次是Illumina测序结果单一数据组装。
表2 测序组装结果统计
3. 注释、直系同源与基因本体
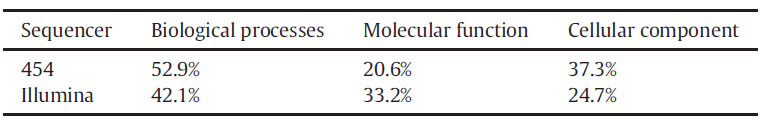
De novo组装非模式生物转录组,采用BLAST注释基因及其功能。本文使用CLC Genomics Workbench软件的BLAST2GO插件,将De novo组装的转录组与其他远亲物种进行BLAST,使用基因本体(Gene Ontology, GO)术语注释基因。总体而言,GO注释三种组装的转录组之间的差异微小,见表3。
表3 GO注释总结
结论:
1.CLC Genomics Workbench软件De novo组装Illumina平台与454平台的单一测序结果与两者的混合测序结果,BLAST注释基因及其功能,BLAST2GO插件使用GO术语注释基因,用于评估De novo组装技术。2.De novo组装结果表明,Illumina测序数据组装结果具有优质的contig长度,454测序数据组装结果具有更完整的基因转录子。混合数据组装和Illumina测序数据组装比454测序数据组装具有更长的contig,转录组的覆盖率更高。混合数据组装因增加了454测序数据比Illumina测序数据组装具有更多的注释基因。
原文:
B. Picone, C. Rhode, R. R. Wilding, Evaluation of de novo assembly technique in the South African abalone Haliotis midae transcriptome: A comparison from Illumina and 454 systems. Genomics Data 10 (2016) 165–166.



