使用免疫亲和蛋白质组学进行PTM肽段的相互作用网络与通路分析
Jeffrey C. Silva1; Stuart Tugendreich2; Hongbo Gu1; Charles L.Farnsworth1; Kimberly A. Lee1; Xiaoying Jia1; Jian Min Ren1; Matthew P. Stokes1
1Cell Signaling Technology, Danvers MA USA01923; 2Ingenuity Systems
Introduction
蛋白质翻译后修饰在细胞信号通路传递中扮演了涵盖蛋白活性调控、稳定及亚细胞定位等方面重要角色。Cell Signaling提供基于抗体技术的翻译后修饰位点(包括磷酸化、泛素化、乙酰化和甲基化等)、共motif激酶底物及来自同一个或多个信号通路的蛋白质肽段的富集方法。这些抗体技术使用免疫亲和纯化富集来自于细胞系、组织、移植物或其他生物材料中的翻译后修饰肽段,然后联用LC-MS/MS技术鉴定数千种以上的翻译后修饰肽段。最后我们采用Ingenuity® Systems 的通路分析软件IPA® 对这些大数据集进行生物信息学的整合与分析。
Methods
使用了Cell Signaling Technology®的Acetyl-Lysine, Mono-Methyl-Arginine, or Ubiquitin Branch 抗体对HCT-116人大肠癌细胞系和小鼠肝脏组织进行了后修饰肽段富集并用PTMScan® 进行位点分析 (Rush et al. 2005, Lee et al. 2011, Kim et al. 2011)。样品分析使用LTQ-Orbitrap®VELOS 或Elite LC-MS/MS,搜库使用Sorcerer (Lundgren et al. 2009)。鉴定结果导入Ingenuity®Systems IPA®软件包进行分析。所有富集得到的翻译后修饰肽段对应的蛋白采用Core analysis进行分析,背景数据库采用Ingenuity Knowledge Base中所有基因和内源性化合物库,所有相互作用分析限制在实验发现或高可信度预测的直接相互作用。
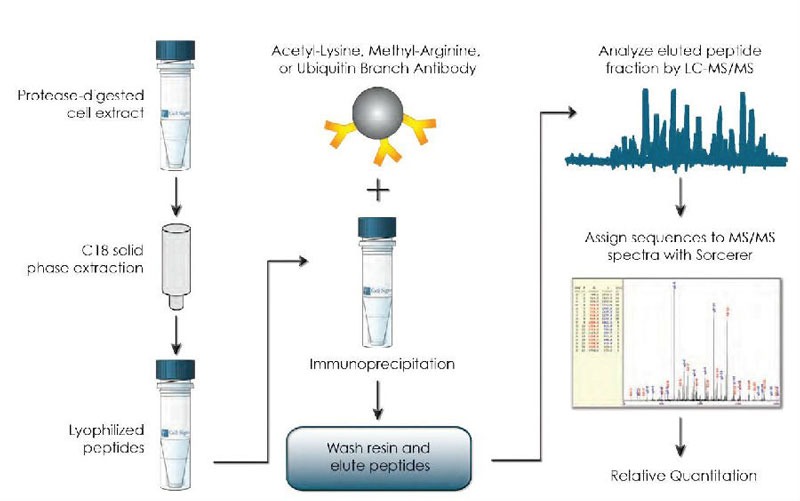
Figure1: PTMScan方法: 免疫亲和纯化实验流程,采用motif特异抗体富集acetylated (AcetylScan®), methylated (MethylScan)和ubiquitinated (UbiScan®) 肽段。
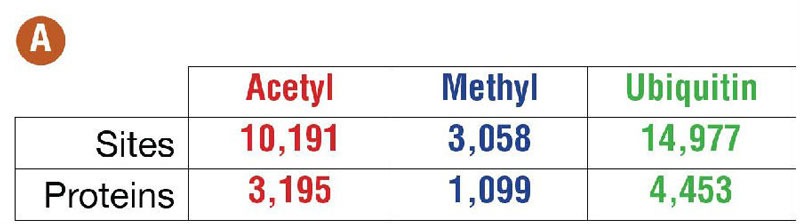
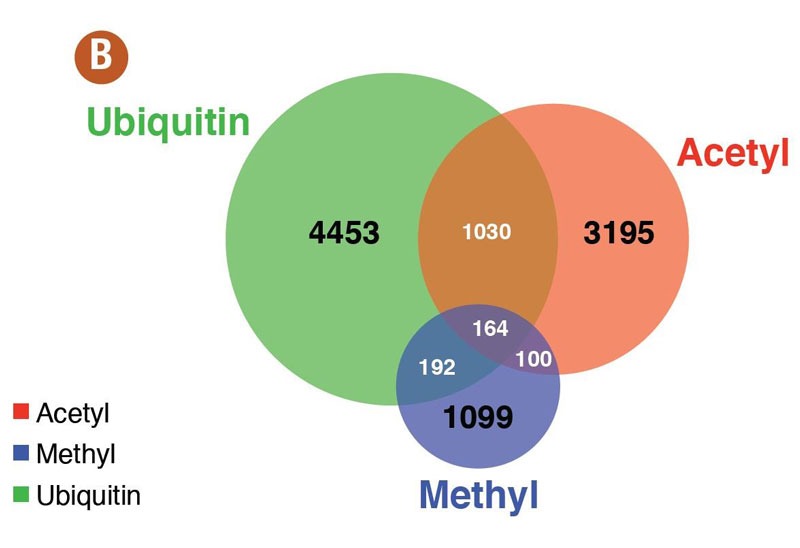
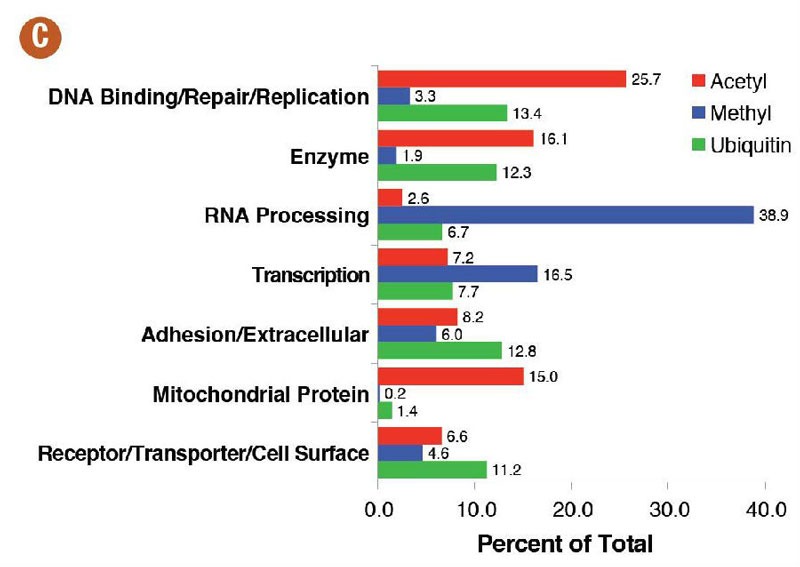
Figure2: 使用Acetyl-lysine, Mono-methyl-Arginine, and Ubiquitin Branch motif抗体进行PTMScan®分析结果.
A. 每类抗体鉴定到的非冗余位点/蛋白数量。B.面积百分比Venn图展示了蛋白鉴定数,每类抗体(黑色数字)和抗体间的交盖度(白色数字)。C. 每类抗体富集蛋白的蛋白类型排名前七的结果。信息来自PhosphoSitePlus®数据库。
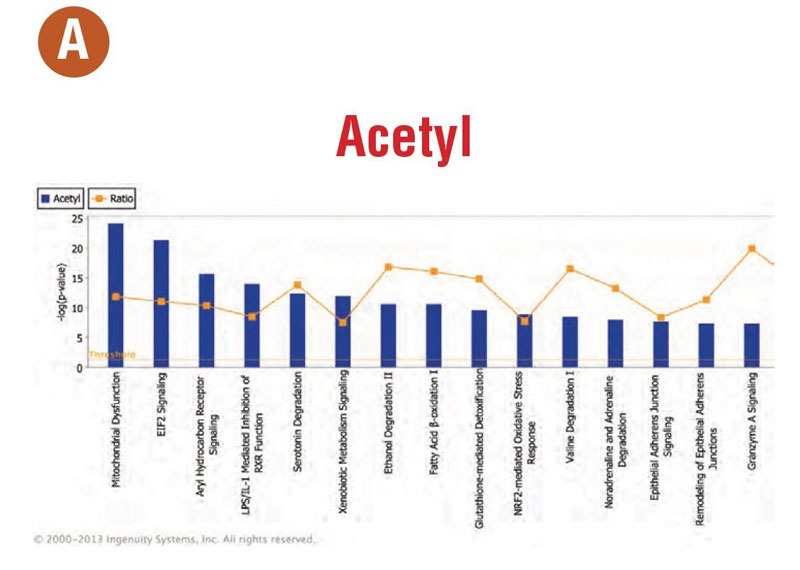
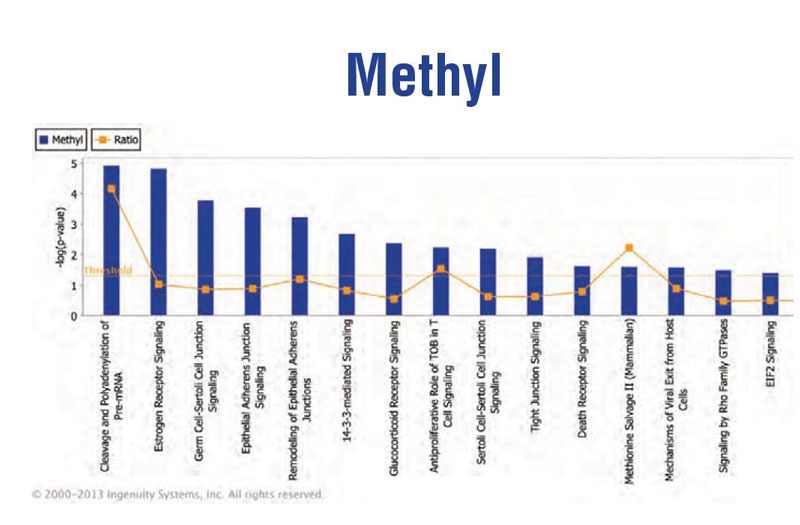
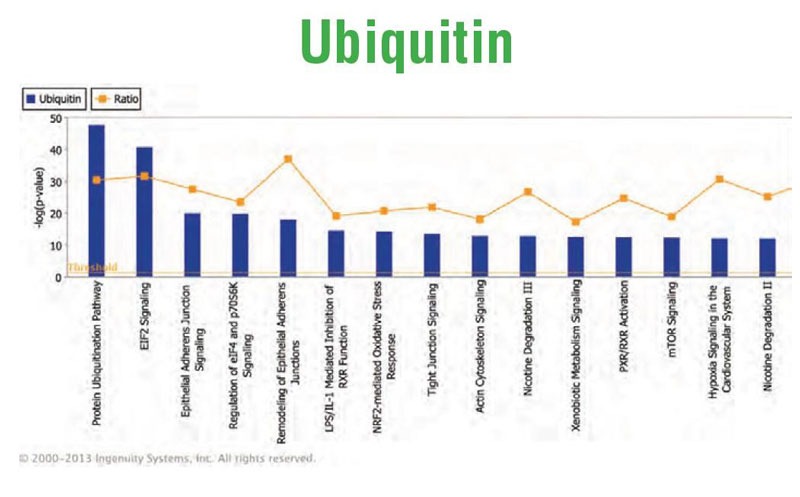
IPA® Canonical Pathways: 将鉴定到的蛋白映射到IPA经典通路数据库
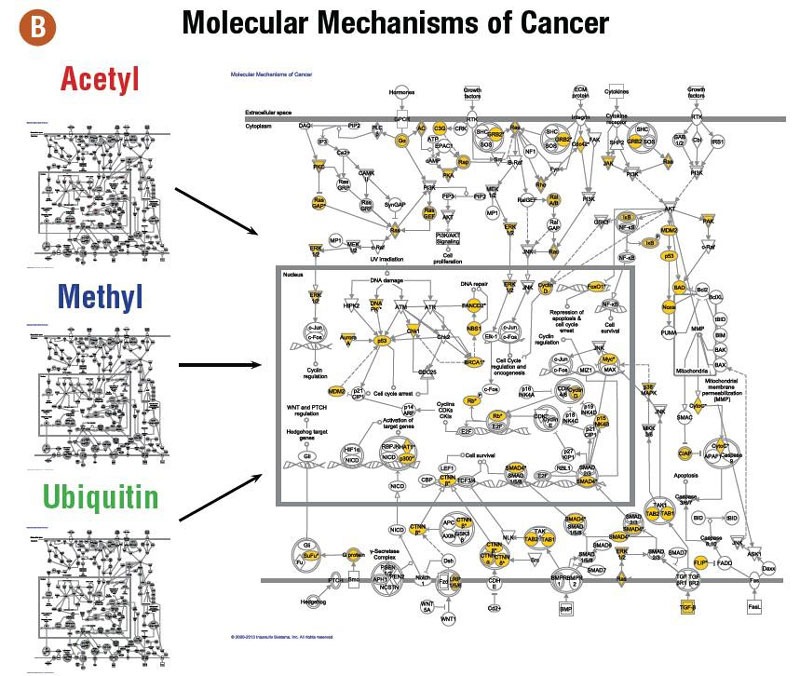
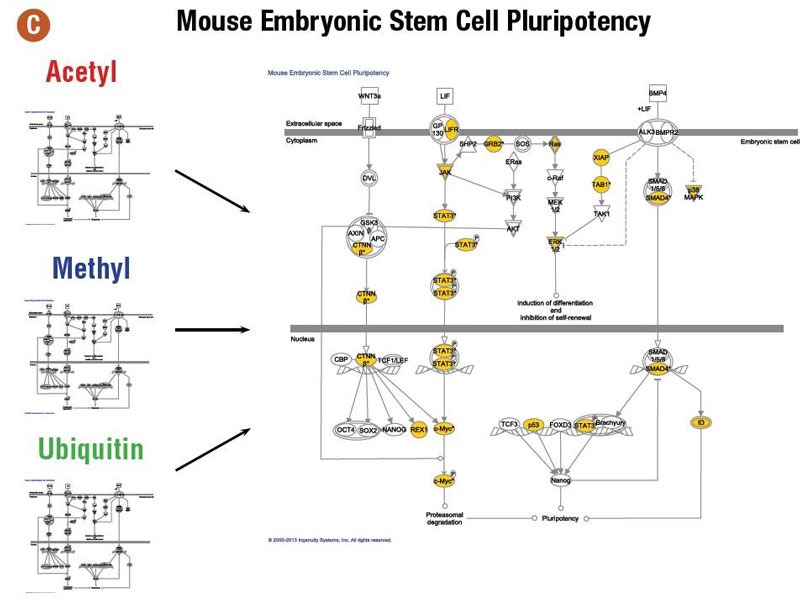
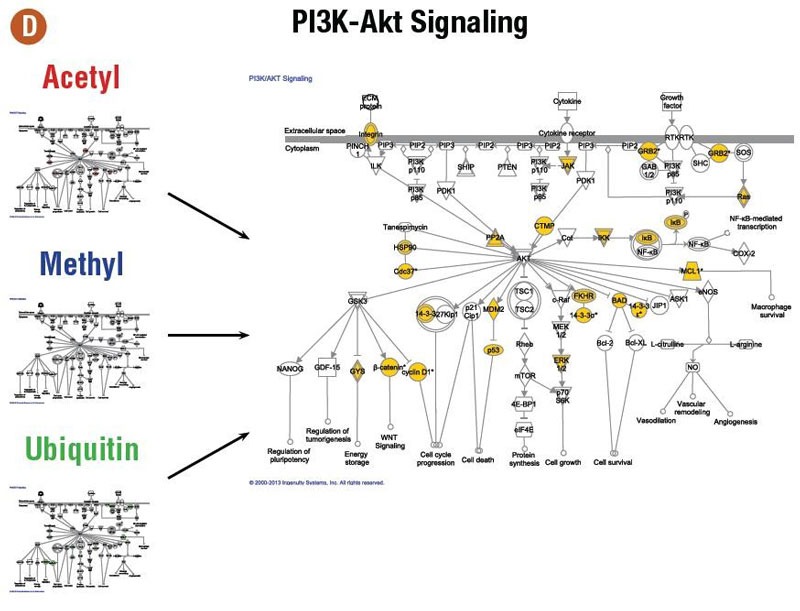
IPA® Network分析: 由鉴定到的蛋白预测的De novo 通路
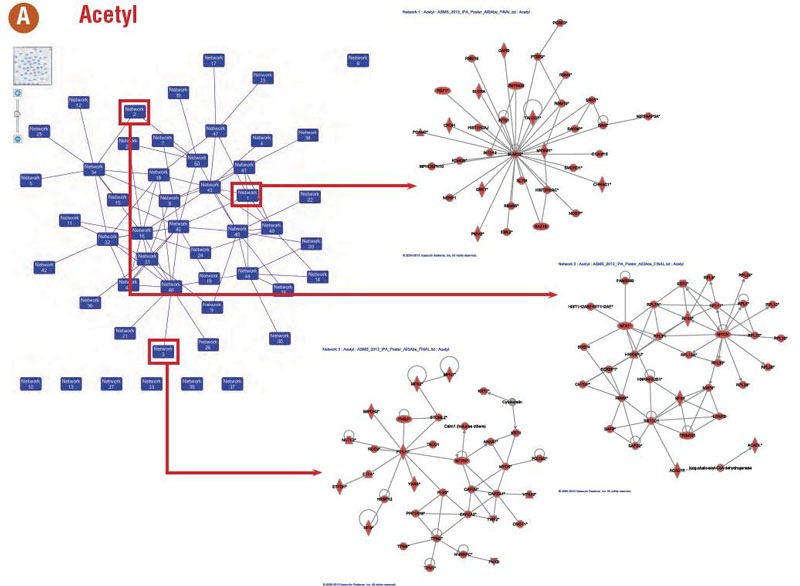
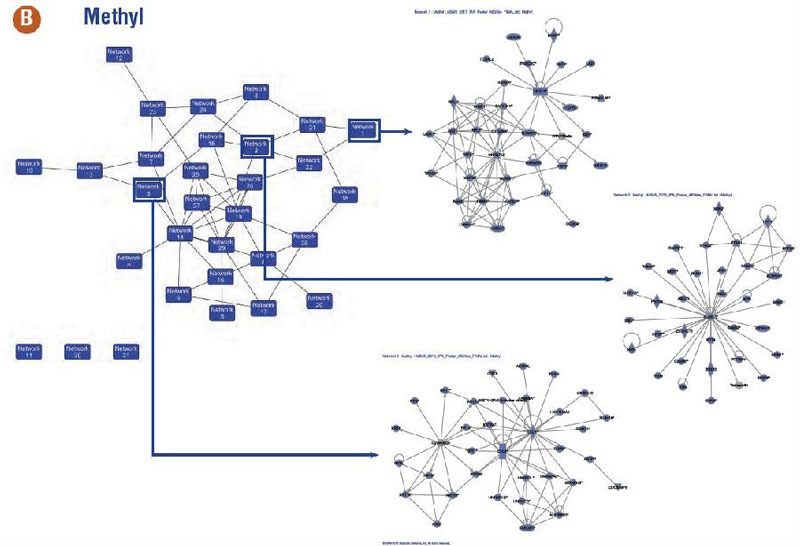
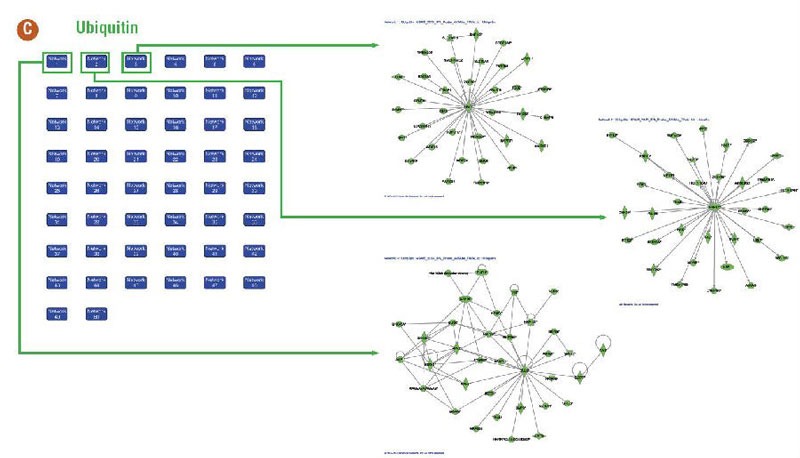
Figure 4: 每类抗体富集蛋白排名前三的IPA®相互作用网络(A =Acetyl, B = Methyl, C =Ubiquitin)。相互作用网络间的共同分子由网络节点间的连线注明。
Conclusion
Ingenuity® Systems IPA® 软件提供了一个深入挖掘由PTMScan®技术抗体富集Acetyl-Lysine,Mono-Methyl-Arginine, and Ubiquitin-Branch motif得到的后修饰蛋白的相互作用网络和通路信息。
References
1.Rush, J. et. al. (2005) Nat. Biotechnol. 23, 94–101.
2.Lee, K. A. et. al. (2011) J. Biol. Chem. 286(48):41530–41538.
3.Kim, W. et. al. (2011) Mol. Cell. 2011 Oct 21;44(2):325–340.
4.Lundgren, D. H. et. al. (2009) Curr. Protoc. Bioinformatics Chapter13, Unit 13 13.



