最新发表的多篇新冠研究文献中使用CLC软件进行数据分析时,多次提及Local realignment工具,可见其优越性得到了认可。本文在解读文献的同时,也将介绍Local realignment工具的原理,旨在帮助大家更好地使用这个工具。
文献:恒河猴在mRNA-1273疫苗接种一年后对SARS-CoV-2 Delta的防御作用与肺中的记忆抗体反应一致(2022)
影响因子:41.58
期刊:Cell
mRNA-1273疫苗对SARS-CoV-2 Delta的效力随着时间的推移而减弱;然而,关于免疫反应持久性对保护的影响数据有限。此研究中研究者对恒河猴进行了免疫,并在1年内评估了血液和上下呼吸道的免疫反应。研究发现,恒河猴在mRNA疫苗接种一年后,肺部存在持久但延迟的记忆抗体反应。上呼吸道的保护有限,中和反应较低,因此提倡加强注射疫苗,以持续保护上下呼吸道。
研究者使用CLC软件(版本21.0.3)分析了demultiplex后的病毒测序数据,步骤包括:
(1)trim,质量控制,
(2)映射到Wuhan-Hu-1 SARS-CoV-2参考基因组(GenBank:MN908947.3),
(3)在包含INDEL的区域通过local realignment工具优化alignment,
(4)生成样本共有序列和变体列表。
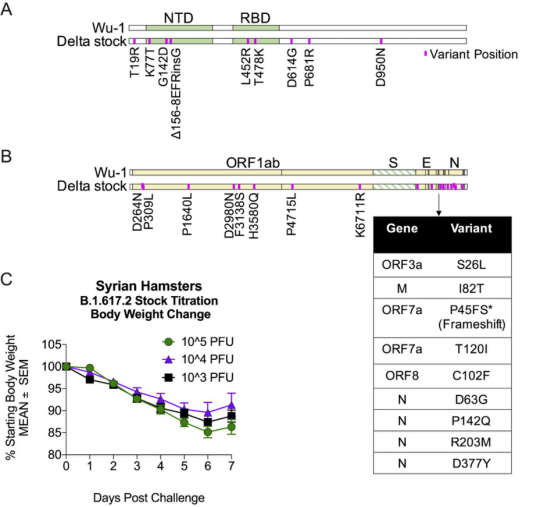
下图A和B展示了B.1.617.2与Wuhan-Hu-1参考序列的alignment。A为只有S基因,B为全基因组。表中显示了基因组3′端的多个突变。
详细介绍Local Realignment工具
Local Realignment工具旨在提升现有read mapping中的序列比对。该算法在尝试重新比对任何特定的read时,会充分利用其他read比对中可用的信息。大多数mapper不使用交叉read信息,因为在mapping算法中在计算层面上这样做是禁止的。然而,一旦read被mapping,Local Realignment就可以利用这些信息。CLC中此工具的算法借鉴了文献[Homer N, 2010]中描述的方法。
算法的第一步,将所有输入的read的比对信息收集在一个高效的可视化数据结构中,该结构本质上类似于de-Brujn图。这个图可以显示read是如何与参考序列比对的,以及read如何相互重叠。在第二步中,从图中派生出元数据来,显示在哪些比对位置重新比对可能会提升read mapping,并且还展示了如何重新比对read可能产生最简洁的多重比对。在第三步中,使用realignment图及其元数据实际执行每个read的Local Realignment。
下图显示了使用Local Realignment前(A)后(B)的比对情况。

在下图中,使用Local Realignment的“global”选项后,绿色的read与插入的“CCCG”序列(从红色read的比对中可以看到)可以map,并且没有出现不匹配。

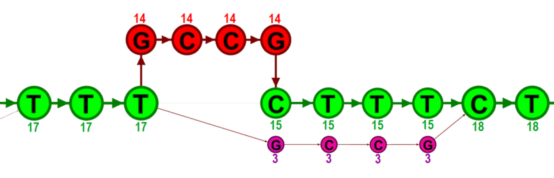
下图举例具体展示了realignment是如何进行的。绿色节点代表参考序列的核苷酸。4个红色节点代表在14个map的read中观察到的4个核苷酸插入。4个紫色节点表示在3个map的read中观察到的参考序列的4个不匹配。在重新比对原始read的过程中,发现了2条可能的路径。一条路径穿过4个红色节点,另一条路径穿过4个紫色节点。由于在14个原始read中看到了红色节点,而在3个原始read中只看到了紫色节点,因此通过4个红色节点的路径比通过紫色节点的路径更可取。
在新冠疫情研究中,NGS技术使得获取病毒基因组变得方便快捷,使用NGS数据对病原生物学特性可进行准确分析,为疫情的防控和疾病的诊断提供依据。测序技术的快速发展对病原检测、疾病研究等带来巨大的技术和应用革新,同时也给相关从业者带来巨大的挑战和发展机遇。CLC软件集成了诸多优秀的算法和数据分析工具,而且其性能也在每次的软件更新中不断提升,相信它会成为您得力的帮手。
[参考文献]
1. Gagne, Matthew, Kizzmekia S. Corbett, Barbara J. Flynn, Kathryn E. Foulds, Danielle A. Wagner, Shayne F. Andrew, John-Paul M. Todd et al. "Protection from SARS-CoV-2 Delta one year after mRNA-1273 vaccination in rhesus macaques coincides with anamnestic antibody response in the lung." Cell (2021).
2. Homer, Nils, and Stanley F. Nelson. "Improved variant discovery through local re-alignment of short-read next-generation sequencing data using SRMA." Genome biology 11, no. 10 (2010): 1-12.



