IPA分析转录组数据,揭示ALV-J病毒在SPF鸡体内的治病性炎症应答机制
背景
禽类白血病病毒(Avian leukosis virus,ALV)是禽类的治病病毒,导致禽类死亡和禽类健康下降,造成经济损失惨重。
864个基因/7个miRNAs/17 IncRNA在感染ALV与没感染ALV的鸟类中差异表达。
实验材料与方法
材料
无病原体的白来航优质鸡(white Leghorn egg-type chicken)长至40天时,实验组注射鸡白血病病毒J亚群(ALV-J) SCDY1,对照组注射PBS。注射后第40天的脾(实验组与对照组各3例)进行转录组测序。分析测序数据,对mRNA、lncRNA与miRNA表达水平分别进分析,发现差异表达基因,即mRNA差异表达基因、miRNA靶标差异表达基因、lncRNA靶标差异表达基因和病毒共表达的差异表达基因。
软件
IPA软件分析四个数据集,mRNA差异表达基因数据集(mRNA数据集)、miRNA靶标差异表达基因数据集(miRNA数据集)、lncRNA靶标差异表达基因数据集(lncRNA数据集)和病毒共表达的差异表达基因数据集(病毒数据集)。
结果
1.IPA通路分析结果
经典通路分析结果显示,注射ALV组具有6个显著性通路(|z-score| > 2),这些通路都与免疫相关,其中5个通路被激活,只有STAT3通路被抑制,见图1a。IPA预测靶基因具有65个激活的上游调控子,几乎所有重要的调控子与天然免疫相关,其中包括IFNs(7个基因)、IRFs(4个基因)和TLRs(2个基因)。此外,IPA下游分析预测与差异表达基因相关的16个功能,其中炎症和免疫应答疾病是显著相关功能,代谢与生殖系统疾病也具有显著性,见图1b。IPA还预测了一个新的分子作用网络,具有三个核心分子,即NF-kB、IRF1和IRF7。这三个分子与病原体识别过程相关,见图1c。
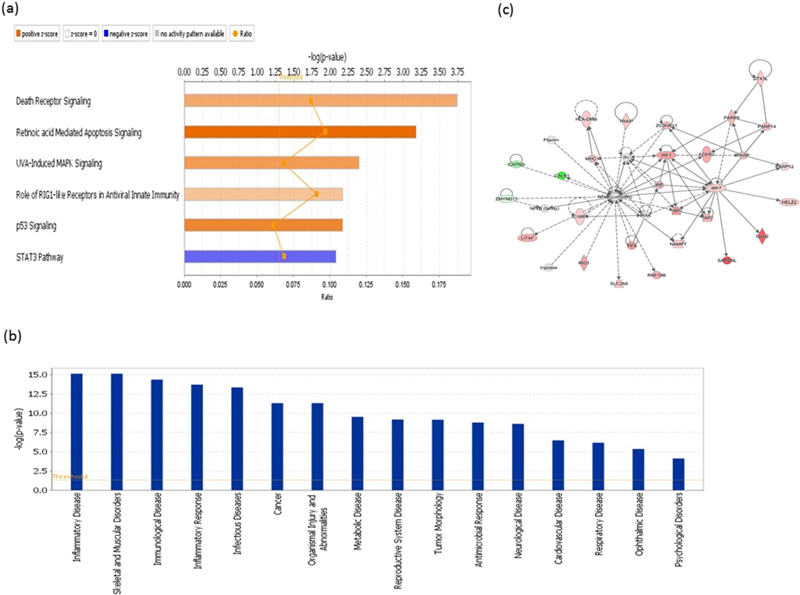
图1 IPA分析与病毒基因相关的差异表达基因 (a)重要的经典通路,橘色表示通路被激活,蓝色表示通路被抑制,颜色越深,表示作用活性越显著。(b)下游疾病和生物通路。(c)预测的分子作用网络显示大量与免疫相关的基因以中心分子NF-κ B、IRF1 和IRF7相关,这三个分子与病原体识别过程相关。点线表示非直接作用关系,实线表示直接作用关系。红色的基因表示表达上调的基因,绿色表示表达下调的基因,灰色表示IPA预测的基因。
IPA分析260个miRNA靶标差异表达基因的数据集,功能分析结果发现其中204个基因与癌症功能相关。经典通路分析结果包括所有与病毒共表达的差异表达基因数据集相关的通路,其他激活的通路是death receptor signaling、 retinoic acidmediated apoptosis 与UVA-induced MAPK biosynthesis I,抑制的通路是cAMP mediated signaling。上游分析预测27个与免疫相关的基因处于激活状态,包括IRF1、 IL5、 IFNG 和 STAT1。IPA功能分析预测lncRNA靶标差异表达基因的数据集,发现390个基因中有254个基因与癌症疾病相关。该数据集经典通路分析结果包括来自病毒组的所有通路,包括被抑制的glioma signaling。
2.IPA对比分析结果
另外,IPA比较分析四种类型数据的综合数据集,分析不同数据之间的同性。四个数据集的几个重要的显著性通路是相同的,见下图2a。前三个显著性通路中有两个通路 “Retinoic acid Mediated Apoptosis Signaling”与“Death Receptor Signaling”被激活,而“cAMP-mediated signaling”被抑制。mRNA数据集经典通路的Z值绝对值最小。有趣的是,几乎所有预测的下游疾病与功能被抑制,包括“lymphoproliferative disorder”、 “migration of cells” 与“vasculogenesis”,见图2b。四个数据集的上游调控因子分析结果具有较好的一致性,见图2c。大多数激活状态的上游调控子与天然免疫相关,例如,IFNA2、 IFNG、 IRF7、 CSF2、 IFN beta 、TLR9、IRF1、TLR3 和STAT1,高度富集到模式识别受体通路与免疫应答。
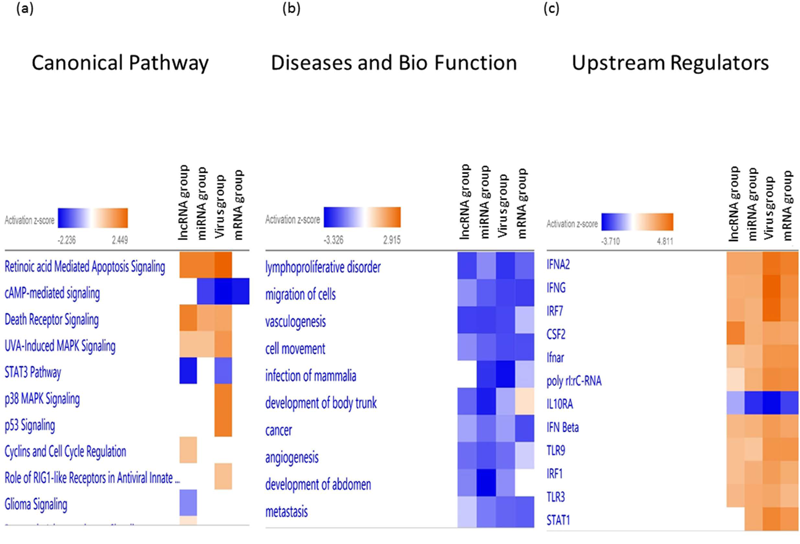
图2 IPA对比分析四个数据集的热图 从左到右:lncRNA数据集,miRNA数据集,病毒数据集和mRNA数据集。(a)重要的经典通路,(b)疾病和生物功能,(c)预测的上游调控子。
3.IPA分析mRNA、 miRNA和lncRNA数据集的共表达网络
研究特定调控的基因与相应调控因子相关,WGCNA用于决定特定基因表达与所有差异性表达的编码蛋白的基因相关,miRNA、lncRNA数据集和病毒数据集。本文发现34个不同的共表达网络,分别具有5-62个基因不等,见图3a。其中4个网络(第1个、第6个、第28个和第29个),这4个网络中的基因与ALC-J刺激和免疫应答相关。
第1个网络包括10个miRNA、1个lncRNA和 27个编码蛋白的基因,FSTL4基因作为中心分子,见图3b。27个编码蛋白的基因中的19个基因与疾病多态性和肿瘤相关。网络的一部分包括3个miRNA——miR-223、miR-6670-5p和miR-190-3p,1个lncRNA——LOC_013679,与14个编码蛋白相互作用,包括Lef1和TAP1。其他7个miRNA——miR-460B-5P、miR-383-5p、miR-181b-5p、gga-let-7b、miR-301b-3p、miR-10a-3p和miR-217-5p,其他13个相互作用的编码蛋白的基因构成网络的另一半,这些分子与免疫系统、关节积血、闭塞性动脉硬化和细胞凋亡等相关。
Novel_48作为一个新miRNA是第6个网络的中心分子,连接两部分编码基因的蛋白,见图3c。这个网络中所有编码蛋白的基因与疾病免疫应答相关,一部分包括HDP基因家族——AvBD1、AvBD4、AvBD6和AvBD7,另一部分12个基因中的10个基因与疾病功能相关,例如,RASGEF1B释放了GDP,激活RAS信号传导。
第28个网络是发现的两个最小网络之一,ALV基因——env与2个miRNA(miR-7 与novel_51)、8个编码蛋白的基因直接作用,见图3d。
lncRNA LOC_009715是第29个网络的中心调控子,与其他9个编码蛋白的基因相互作用,见图3e。
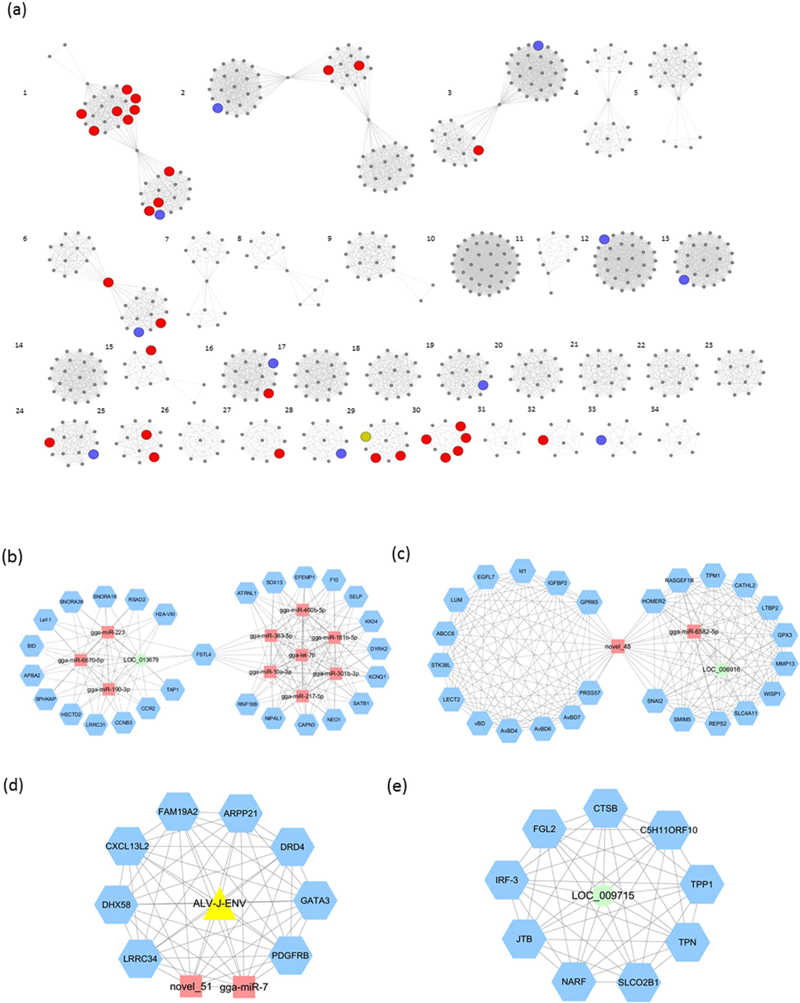
图3 四个数据集的共表达网络分析结果 (a)网络分析发现34个网络,每个网络至少具有7个基因。(b)网络1包括10 个miRNA、1个lncRNA和27个编码蛋白的基因。(c)网络6具有一个新miRNA——novel_48,作为这个网络的中心基因,连接2部分编码蛋白的基因。(d)网络28具有ALV基因-env,直接与2个miRNA——miR-7 和novel_51、8个编码蛋白的基因相互作用。(e)网络29具有一个lncRNA——LOC_009715作为这个网络的中心调控子,调控9个编码蛋白的基因。
结论
1.IPA对来自实验组的380个差异性表达的基因的分析结果显示,宿主对病原体的反应机制包括病原体识别和抵御,细胞凋亡和免疫通路被激活。
2.经典通路分析发现了p53 signaling、RIG-I 和STAT3通路。
3.下游功能分析预测一系列免疫应答
4.网络分析发现了3个关键基因——NF-κ B、IRF1和IRF7,它们与7个表达上调的基因相互作用,这些基因与多种癌症相关。



