澳大利亚的研究者(Donner, et al., 2021)发现,废水检测可在新冠病毒感染者出现症状几周前就发现社区中存在的新冠病毒,有助于构建有针对性的早期检测系统。研究人员还表示,如果采样足够频繁和广泛,该技术可在有患者感到不适前通过废水检测出新冠病毒,这是因为他们在得知自己被感染前,身体已开始通过粪便将病毒碎片排入废水系统中。
一些患者可能在表现出临床体征前就已感染并传播新冠病毒。有证据表明,新冠病毒感染人体后,可在患者出现症状前的一段时间自我复制,一部分病毒便会随人体粪便排出体外。这一发现有助于针对特定区域进行防疫措施限制,避免较大区域的“完全封锁”,有利于恢复经济和让人们更自由流动。废水的微生物分析可以对病毒传播起到定期监测和优质预警的作用。对废水进行基因分型测序可以有效监测集水区内的病毒流行病学,废水样品随时可用,收集便捷。避免医院或检测机构采样的偏差。
本文将介绍如何通过CLC软件批量下载感兴趣的公共数据集,并通过workflow进行分析,导出consensus sequence并上传到Pangolin进行分型确认。
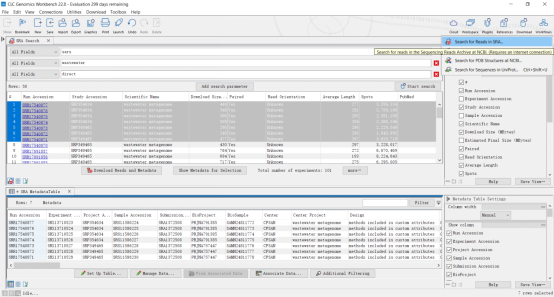
1. 首先,从CLC软件右上角的download搜索关键字并下载相关的数据集,如下所示为SRA数据库下载的界面,可以添加多个关键字进行搜索,下部的metadata表格中包含采样地点、试剂盒类型等信息。
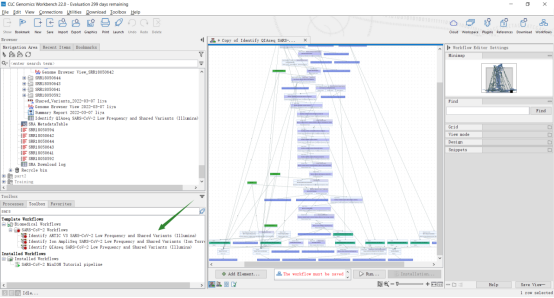
2. 从toolbox中选择workflow用于分析,下图左下角显示了3个SARS-CoV 2的模板workflow,以及一个单独导入的workflow。
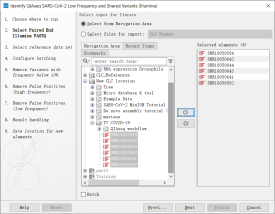
3. 选择需要分析的文件,设置参数并运行。
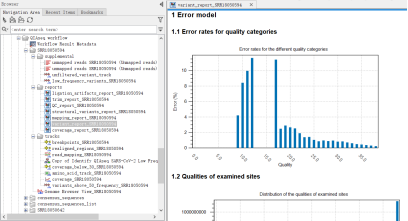
4. 获得workflow分析的结果
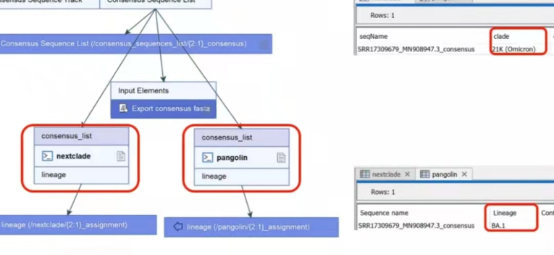
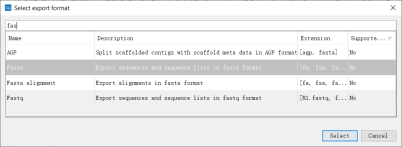
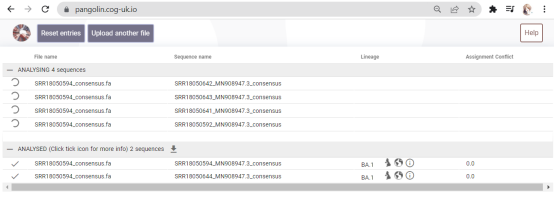
5. 从CLC导出生成的consensus sequence文件(fasta),并导入pangolin网站进行分析,确认毒株信息。
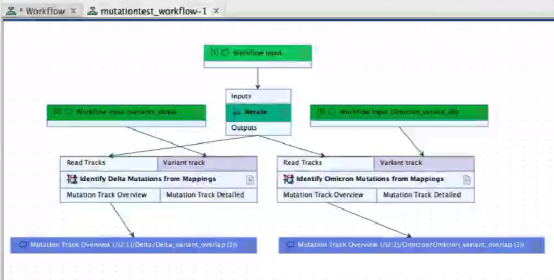
6. 构建进行变异测试的迭代workflow。