

IPA(Ingenuity Pathway Analysis)软件常见客户提问集锦
问题1:初次使用如何登陆?
回复:
IPA登录说明如下:
(1)客户端IPA-China下载地址:https://analysis.qiagenbioinformatics.cn/pa/installer/select,Mac系统、Windows系统选择不同客户端。
(2)客户端安装及登录
IPA客户端安装在电脑后,桌面显示IPA的图标 。双击,打开IPA软件。
。双击,打开IPA软件。

图1-1 打开IPA软件,缓冲过程如上图所示
在弹出的登录界面中输入申请IPA账号使用的Email及密码,即可成功登录,进入IPA操作界面,如下图所示。
图1-2 IPA软件界面
问题2:收到原始密码后,自己如何设置密码?
回复:打开链接:https://apps.qiagenbioinformatics.cn/isa/account/forgotpassword?start=
请勿直接点击软件forget password,因为跳出来的链接是IPA US instance的密码修改界面。在下图界面Email框中输入IPA软件的登陆邮箱地址,点击Submit。待收到Qiagen公司提供的重置密码链接,重置密码即可。(以上操作请在注册IPA软件的电脑上完成)
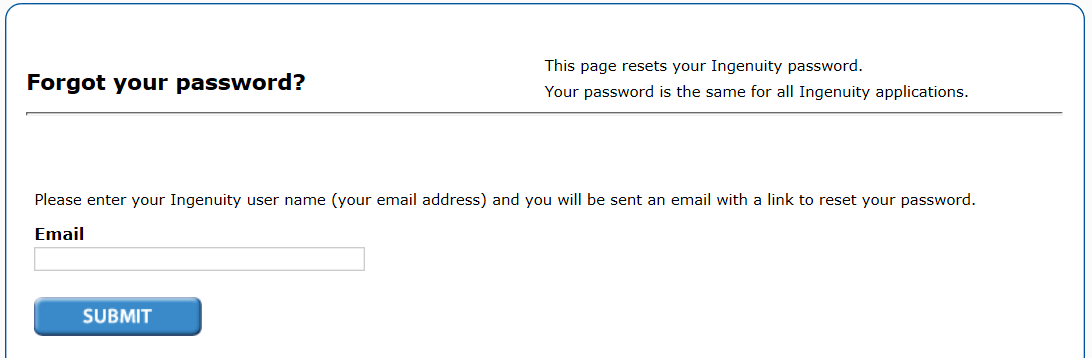
图2-1 申请重置密码界面
问题3:IPA分析数据的数据格式?对数据类型与数值范围的要求是什么?
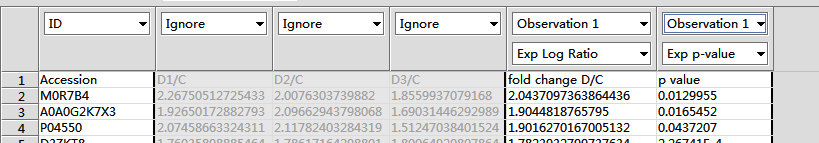
回复:IPA可以识别 .txt、.xlsx和.xls 格式的文件,推荐使用Excel第一张表格编辑数据,表格中一定要包括ID(即表格第一列是基因、蛋白或化合物的ID)和实验数据值,每列只有一个抬头。每个表格中可以最多包括20组观察(Observation),观察结果最多对应3种数值类型。如图3-1所示。IPA能够识别的数值类型与数值范围,见表3-1。
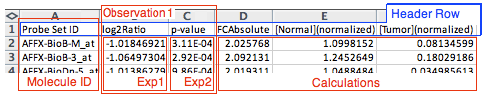
图3-1 Excel编辑数据格式示例
表3-1 IPA能够识别的数值类型及其有效数值范围
表达数据类型 | 有效数值范围 |
Ratio | (0, +INF) |
Fold Change | (-INF, -1) (1, +INF) |
LogRatio | (-INF, +INF) |
p-value | (0, 1) |
FDR, q-value | (0, 100) |
Intensity | (0, +INF) |
RPKM/FPKM | (0, +INF) |
Other (normalized around zero) | (-INF, +INF) |
Variant Loss/Gain* | (-2, -1, 0,1, 2) |
Variant ACMG Classification | (-2, -1, 0,1, 2) |
问题4:代谢物的ID我们一般都是用HMDB的数据,如这个肾上腺素Epinephrine,我在excel中输入HMDB00068还是00068即可?
回复:输入Epinephrine在HGMD数据库中的ID:HMDB00068。IPA识别的数据库及对应ID见表4-1,供参考。
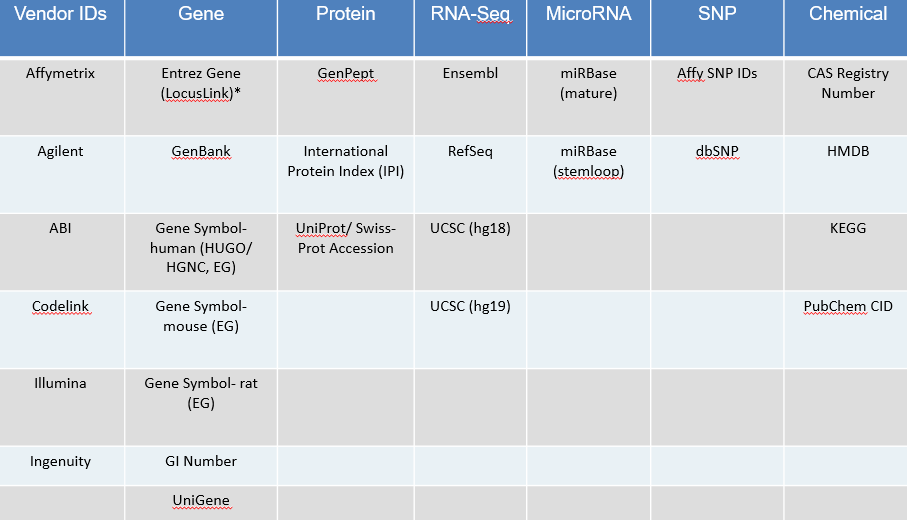
表4-1 IPA识别的数据库
问题5:尝试上传数据到IPA,在最后一步弹出对话框,这个有影响吗?
回复:这个对话框是提示您可以填入一些关于数据集的元数据,比如所上传数据的样本来源、患者的基本背景信息、样本处理方式等信息。这些信息可方便之后对数据进行搜索和汇总管理。如果不填写,不影响接下来的分析。
问题6:如何筛选与疾病相关的基因,标准是什么?
回复:快速筛选与某种疾病相关的基因,推荐使用IPA的高级分析模块——BioProfiler。标准依据用户感兴趣的内容而定。使用BioProfiler筛选与疾病相关的基因,操作步骤如下:
(1)以疾病Vesicoureteral Reflux(膀胱输尿管反流)为例,在Diseases and Functions搜索栏,搜索Vesicoureteral Reflux,搜索结果见图6-1。在左边勾选框中选择感兴趣的与肾和泌尿疾病相关的分子,使用BioProfiler(点击红色箭头所指处)迅速筛选感兴趣的分子。

图6-1 与疾病Vesicoureteral Reflux相关的分子
(2)分子导入BioProfiler后,显示结果见图6-2。用户可以自定义表格显示的信息,可以在Molecule Type中选择基因(图6-2(1)的下图),还可以根据基因表达的蛋白类型,进一步更准确地选择感兴趣基因。用户还可以根据图6-2所显示的信息类型:组织或细胞系、分子活性、对疾病的影响和突变信息等,进一步筛选感兴趣的基因。
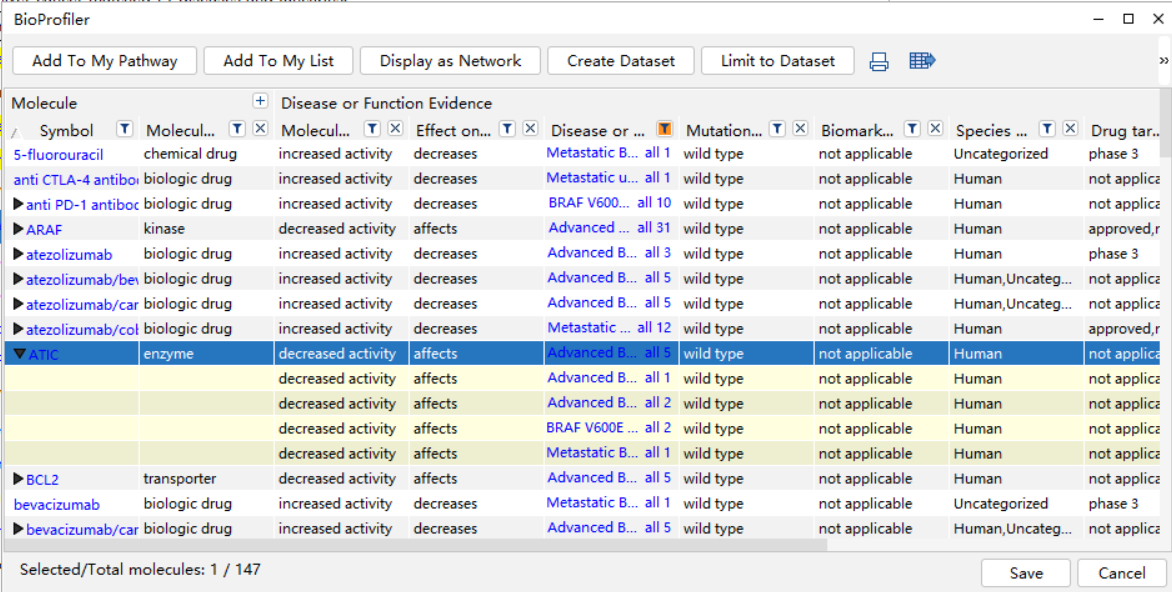
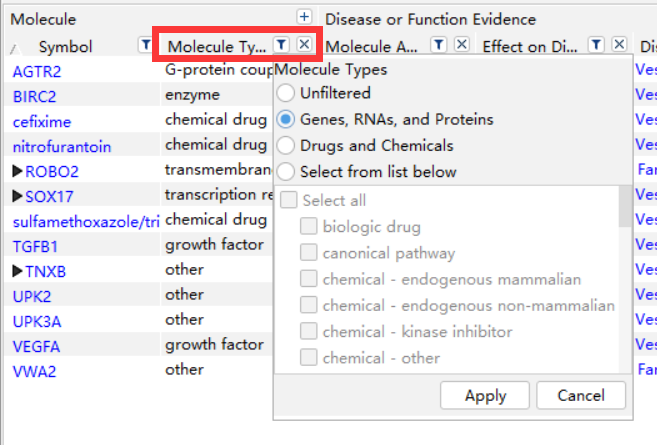
图6-2(1)分子详细信息
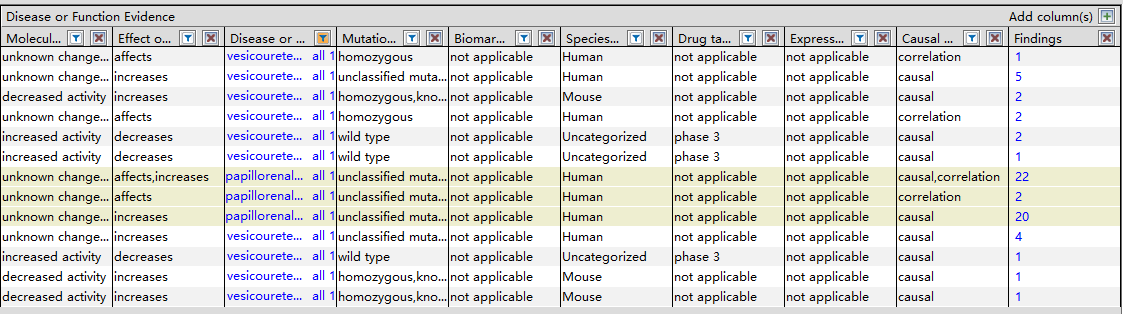
图6-2(2)疾病相关信息
图6-2 将所选分子导入BioProfiler,显示相关的详细信息
问题7:经典通路是怎么得来的?是否可信?
回复: IPA能够查询经典通路信息,提供通路图示(Pathway)与通路文字介绍(Report),信息可溯源。这些信息来自Ingenuity Knowledge Base。Ingenuity Knowledge Base是由多位专业领域的博士或至少具有博士学位的科学家,通过人工阅读从专业文献中提取信息组成的,这些信息经多轮质控,符合Ingenuity Knowledge Base的收录标准。
以ATM signaling为例,点击Pathways and Tox Lists进行搜索,如图7-1。打开ATM signaling图示,如图7-2。如果您还想进一步查看ATM Signaling相关信息的支持文献,请在打开的Canonical Pathway窗口图形区域单击鼠标右键,在弹出的工具选项中选择最后一项View References,便可查看该pathway相关的文献链接和引用信息,如图7-3所示,点击链接即可查看支持ATM signaling的文献。
图7-1 ATM signaling搜索结果
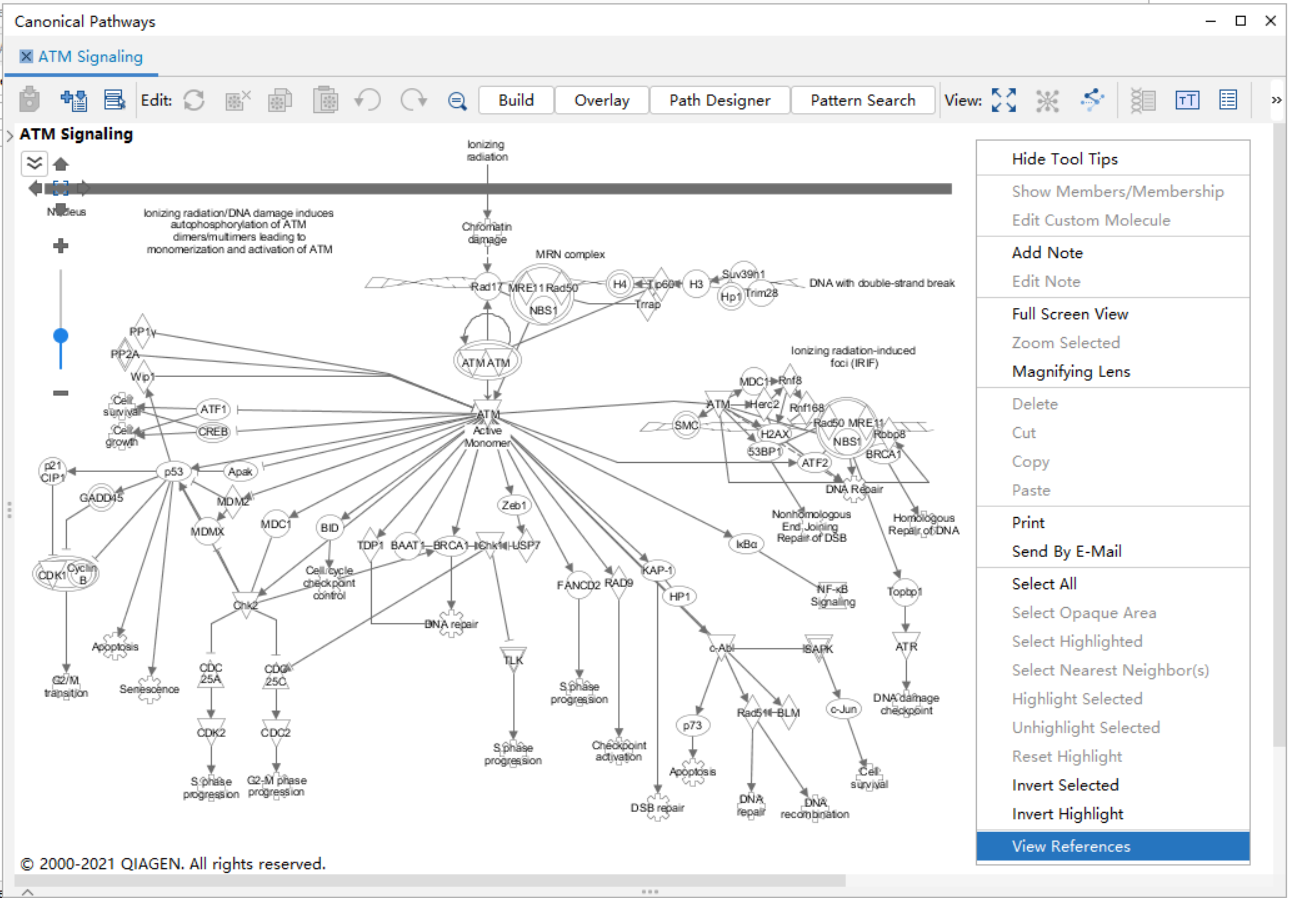
图7-2 ATM signaling图形展示形式
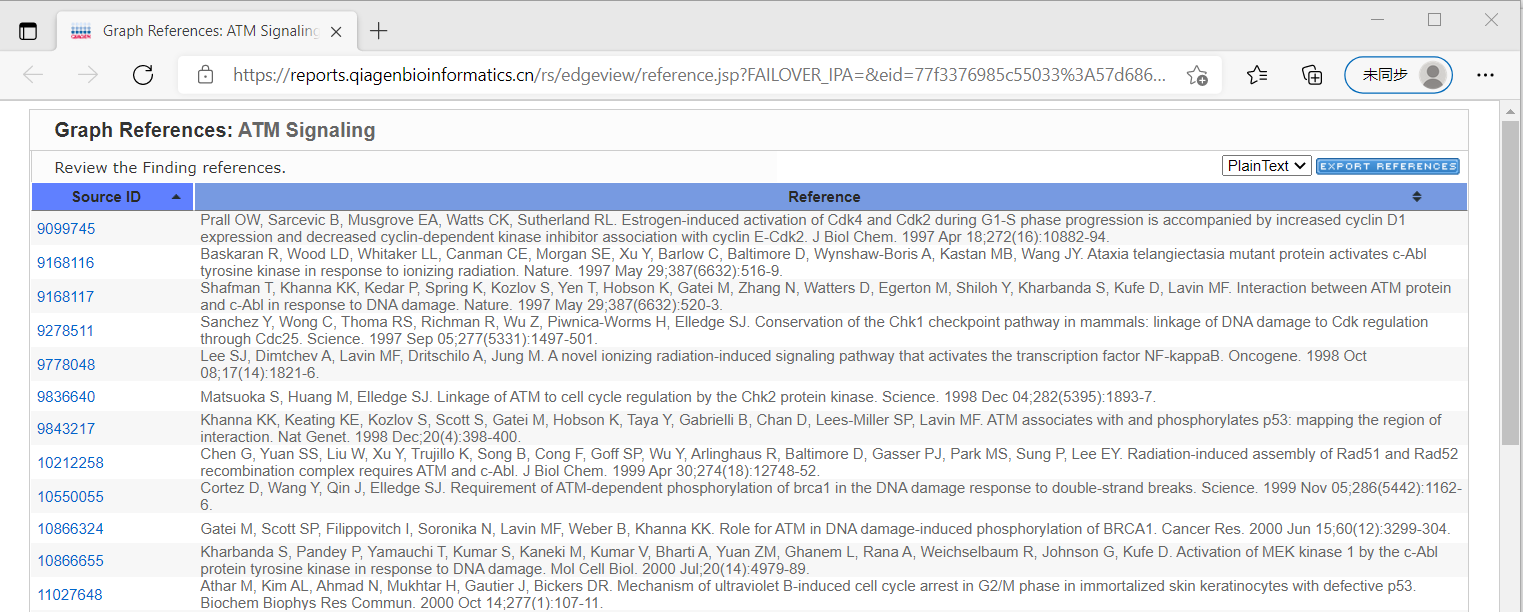
图7-3 支持ATM signaling的文献链接
问题8:如何查询疾病相关药物信息?
回复:(1)在搜索栏中输入疾病名称,例如,Liver Cancer。(2)选择所有搜索到的分子,导入BioProfiler。(3)在BioProfiler窗口,将Molecule Type限定为:drugs and chemicals。筛选结果既包括药物又包括化学物质,不过您可以在Molecule Type栏中指定chemical reagent或chemical drug来显示您感兴趣的分子类型,您可以方便地从结果中区分哪些是药物哪些是化合物,如图8-1所示。
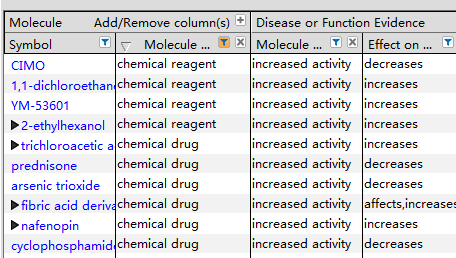
图8-1 在BioProfiler模块中,筛选与Liver Cancer相关的药物或化合物。
问题9:如何查询某种疾病的致病机制?
回复:查询致病机制一般需要限制基因、蛋白等分子类型,如图9-1所示,且以具有对应分子实验数据为佳。原因有二,第一,与某一种疾病相关的分子太多,会搜索出太多可能的致病机制,从中挑选最符合您要求的致病机制并非易事;第二,如果您限定某些分子和某种背景,如:上传某些分子的实验数据,IPA就能够帮您建立一个合理的致病机制假说。
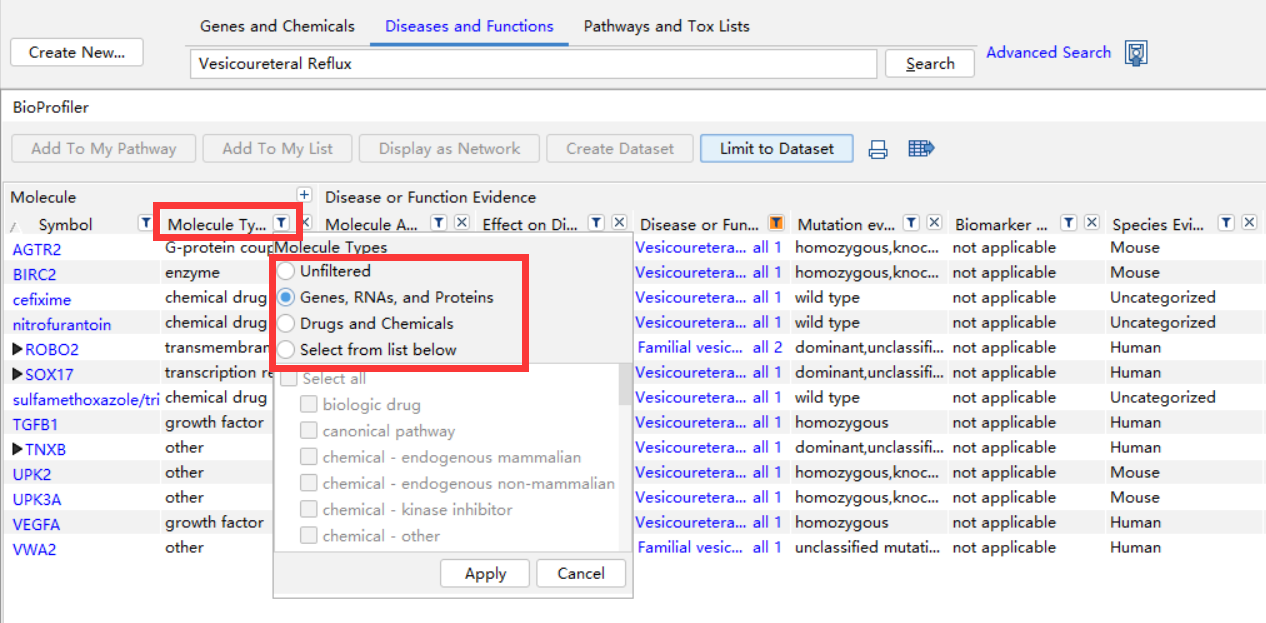
图9-1 在疾病查询模块中,限制基因、蛋白等分子类型
问题10:我想整合代谢组和蛋白组数据进行一体化的关联分析,怎么用IPA进行操作(蛋白的名称和代谢物的名称我都有)?
回复:对于多组学数据的分析,在编辑数据集时可以将不同类型数据ID,如:蛋白ID与代谢物ID放在同一列,但需保证这些ID能够被IPA识别。然后再根据实际需要,选择分析功能。如:代谢组数据和蛋白组数据的整合数据集可以使用Core Analysis进行分析。建议使用蛋白ID或代谢物ID作为IPA识别分子的依据,而不是蛋白名称或代谢物名称。
问题11:我的TTEST值应该选在observation1下的哪个选项?

回复:IPA不支持TTEST值的分析,请查看问题3:IPA支持的数值类型。
问题12:导入数据时,如果选Log Ratio就会出现全部是上调的红箭头;Top Analysis-Ready Molecules分析中,Exp Log Ratio 显示NO data,为什么?
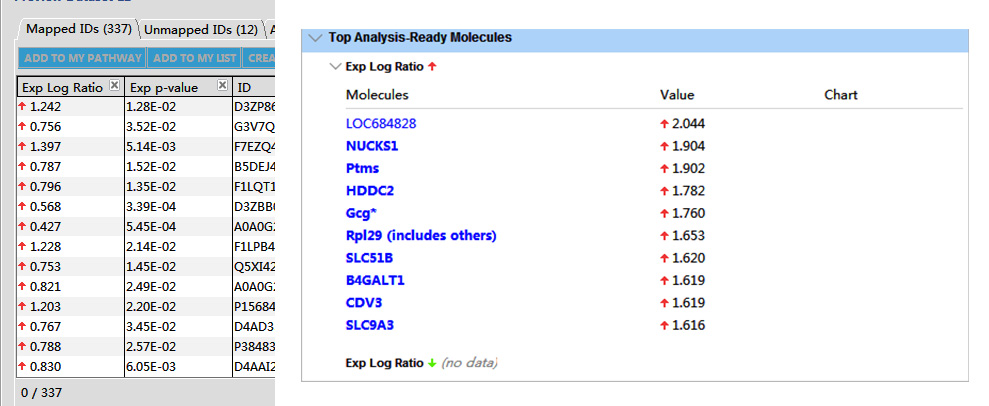
回复:IPA显示的Exp Log Ratio是 ↑ 还是 ↓ 与所上传的数据数值有关,Log Ratio值大于0显示为 ↑ ,小于0显示为 ↓ 。Exp Log Ratio 显示 ↓ NO data是因为上传的数据集中没有小于0的数值。
问题13:如何从经典通路分析中挑选有用信息?
回复:首先要正确解读分析的经典通路结果,用户可从中挑选感兴趣的内容或与研究最相关的通路。接下来以人类肝细胞癌转录组测序(HCC_488)数据集经典通路分析结果为例,进行解读结果解读:
经典通路分析结果显示的内容是与整个数据集相关性显著的经典通路,代表显著性的数值P-value由Fisher's exact test right-tailed方法计算。显著性表示数据集中的分子与经典通路相关的可能性。详细内容如图13-1所示。
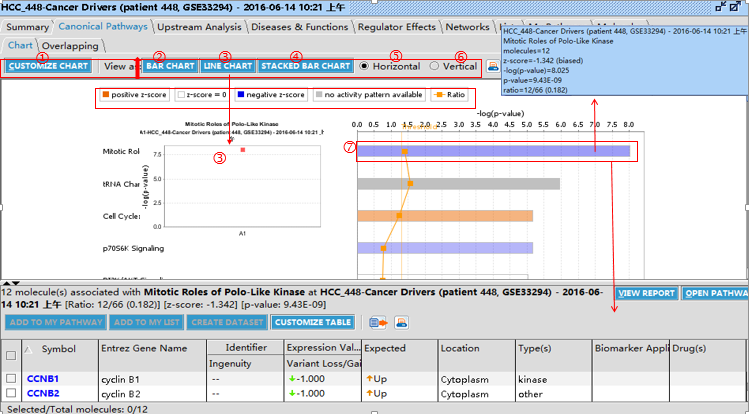
图13-1人类肝细胞癌转录组测序(HCC_488)数据集经典通路分析结果(条形柱呈现方式)
经典通路条形图(图13-1 左图)X轴表示经典通路名称,Y轴表示-log (p-value);条形柱高低表示显著性大小;通路活性状态由不同颜色表示,根据z-score预测通路是激活还是抑制。此分析可快速鉴定数据集中基因差异性表达对通路活性的影响。橘色表示激活,蓝色表示抑制,灰色表示无法预测通路状态,白色表示z-score等于或非常接近0;橘色小方块表示基因数的比值,即数据集中涉及某经典通路的基因数/某经典通路涉及的总基因数。图中-标示内容解释如下。
:定制表参数设置,对应内容见图13-2。
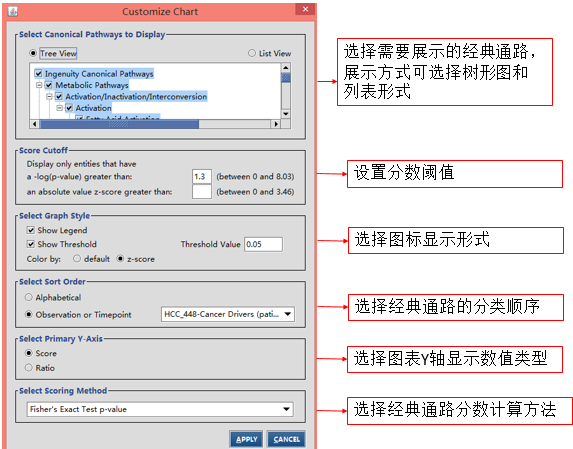
图13-2定制表参数设置界面
 :经典通路3种不同的显示方式。
:经典通路3种不同的显示方式。
 显示条形图形式的经典通路,如图13-1左图所示。X轴表示经典通路名称,Y轴表示 -log (p-value)。显示方式可选择水平显示方式■
显示条形图形式的经典通路,如图13-1左图所示。X轴表示经典通路名称,Y轴表示 -log (p-value)。显示方式可选择水平显示方式■  。●
。●  显示图示见图13-3红色向下箭头指向的蓝色框中内容。○
显示图示见图13-3红色向下箭头指向的蓝色框中内容。○  显示的经典通路图示见图13-3。
显示的经典通路图示见图13-3。
也可选择垂直显示方式□  以垂直方向显示经典通路的条形图如图13-3。
以垂直方向显示经典通路的条形图如图13-3。
□ 显示与HCC_488数据集相关性显著的经典通路是“Mitotic Roles of Polo-Like Kinase”。将鼠标悬停在“Mitotic Roles of Polo-Like Kinase”条形柱上,即可显示数据集中与这个通路相关的基因数、-log (p-value)、z-score、基因数比值。如图13-1红色向上箭头指向的蓝色框中内容。单击“Mitotic Roles of Polo-Like Kinase”条形柱,在下方(图13-1)可显示所分析数据集中参与此通路基因的详细信息列表,其中“Expected”栏所显示的up表示此通路被激活,这些信息有助于预测基因的状态。比如,图13-1中所示,如果通路“Mitotic Roles of Polo-Like Kinase”被抑制,则可以预测基因CCNB1被上调。
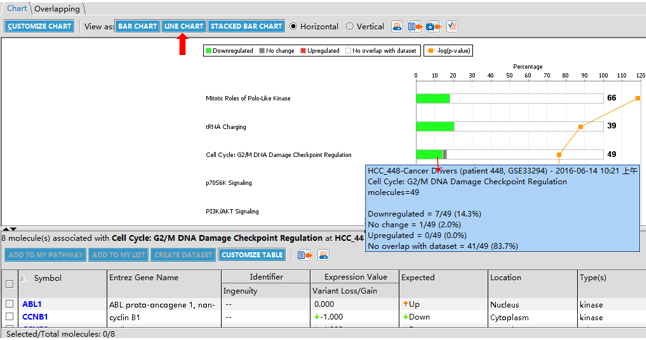
图13-3人类肝细胞癌转录组测序(HCC_488)数据集经典通路分析结果(条形柱呈现方式)
图13-3显示HCC_488数据集中涉及每个经典通路的基因是上调(红色)、下调(绿色)还是无变化(灰色)。橘色点表示每个通路的-log (p-value)。将鼠标悬停在“Cell Cycle: G2/M DNA Damage Checkpoint Regulation”条形柱上即可显示该通路的详细信息,如图13-3中红色向下箭头指向的蓝色框内容。单击“Cell Cycle: G2/M DNA Damage Checkpoint Regulation”条形柱,即可将HCC_488数据集中涉及此通路的8个基因的详细信息显示在图下方的列表中。
问题14:IPA能够修改上传的数据数值吗?IPA能够计算Fold Change吗?
回复:IPA不能修改上传数据的数值,只能选择用或忽略某些数值,或者通过设置阈值选择符合条件的数值用于分析。IPA不是统计学工具,数据上传至IPA之前,请自行做好生物学统计。
问题15:IPA查询次数受限?
回复:IPA不限制查询次数,如果发生这种情况,很可能是您的账号因导出图片次数超过本月上限而被停用(因为IPA对导出图片次数有限制)。请您联系源资公司反馈情况,我们会协助解决问题。
为避免此类情况发生,请用户先将感兴趣的分子网络图或通路图存放在My Pathways中,或使用其他方式截图保存,待需要高清图时再导出,一定时间段内合理分配导出图片的数量。